shotshop.com
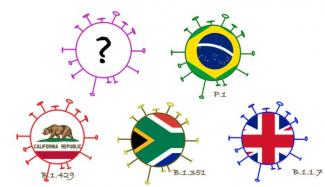
Variants britannique, sud-africain, brésilien : les mutations du coronavirus dominent l'actualité. Que se cache-t-il exactement derrière les différents variants ?
On sait que les virus peuvent muter. Les virus à ARNm comme le SARS-CoV-2 sont particulièrement sujets aux mutations. Depuis le début de la pandémie, on a recensé bien plus de 12 000 mutations. Nombre d'entre elles n’ont pas eu de répercussions. Certaines ont toutefois modifié le virus et donné naissance à un certain nombre de nouveaux variants. Ces variants ont été examinés dès le début de la pandémie. Au Luxembourg, c’est au LNS qu’incombe cette tâche. Au début, il était possible d'observer les différents variants et de retracer plus ou moins l'endroit d’où le virus avait été importé (au début, il y avait donc les variants chinois, italien, espagnol, etc.). Pendant longtemps, il ne s’agissait pas d’un grand sujet de discussion, car les divers variants ne différaient pas substantiellement en termes de potentiel infectieux et de virulence (capacité à rendre les gens malades). Les chercheurs ont cependant signalé dès le début de la pandémie que des mutations plus contagieuses et plus virulentes pouvaient apparaître. Et c'est ce qui s’est produit. On sait à tout le moins que des mutations plus contagieuses sont en train de s'imposer. Il n'est pas encore tout à fait clair si elles sont également plus virulentes, mais certains éléments le laissent entendre. Une mutation virale moins contagieuse ou moins virulente aurait aussi pu s’imposer – comme c'était le cas avec le SARS-CoV-1 (le virus responsable de l'épidémie de SARS en 2002). Cependant, dans le cas du SARS-CoV-2, la situation semble évoluer dans l'autre sens, du moins à l'heure actuelle.
Nous allons à présent analyser plus en détail les différentes mutations :
Infobox
Sur le plan biologique, les virus sont en principe constitués de matériel génétique pur entouré de lipides et de protéines (comme la protéine Spike dans le cas du coronavirus). Le matériel génétique des virus est constitué de molécules d'acide nucléique formant une notice des modes de synthèse des différentes protéines. Contrairement aux êtres humains, aux animaux, aux plantes ou aux bactéries, les virus ne sont toutefois pas en mesure de se servir de ce manuel de synthèse pour fabriquer eux-mêmes les protéines nécessaires à leur réplication. Pour ce faire, ils ont besoin d'une cellule hôte. Une fois qu'ils en ont trouvé une, ils la forcent à lire la notice de synthèse du virus et à produire en boucle de nouvelles copies du virus.
Ces processus de réplication ne se déroulent toutefois pas toujours sans faute. De temps à autre, une erreur se produit et altère le matériel génétique et donne naissance à une mutation. Certaines de ces erreurs de réplication n'ont pas d’effet. D'autres, en revanche, modifient la notice de synthèse de telle manière que de nouvelles protéines se forment, ce qui a des répercussions sur le virus. Il peut devenir plus contagieux ou plus virulent, causant plus de dommages à son hôte. Il peut mieux échapper au système immunitaire, mais il peut aussi être toléré par l’hôte. Les mutations en tant que telles sont des phénomènes aléatoires. Il n'y a pas de stratégie ou d'axe de développement qui les sous-tendent. Du point de vue de l’évolution, les mutations qui favorisent la multiplication et la propagation du virus s’imposent toutefois. Avec le temps, des variants du virus apparaissent, qui peuvent à leur tour former de nouvelles souches.
L'acide désoxyribonucléique (ADN) et l'acide ribonucléique (ARN) sont les molécules de la vie. Tous deux comportent les notices de synthèse de toutes les protéines qui forment l'organisme, et les transmettent à la génération suivante. Les notices de synthèse sont rédigées à l’aide d’un alphabet constitué de quatre lettres seulement, à savoir les bases adénine, cytosine, guanine et thymine. Dans l'ARN, l'uracile remplace la thymine. Alors que les informations génétiques chez l’être humain et les animaux, les plantes et les champignons, ainsi que les bactéries et les archées sont stockées dans l'ADN, chez certains virus – dont le SARS-CoV-2 – elles sont stockées dans l'ARN.
La séquence des bases dans l'ADN et l'ARN constitue le code pour synthétiser les protéines. La notice de synthèse du SARS-CoV-2, par exemple, compte environ 30 000 lettres. Celles-ci doivent être transcrites chaque fois que le virus se réplique dans la cellule hôte. Des fautes d'orthographe se produisent pendant ce processus. Il arrive qu'une lettre soit oubliée (deletion). Parfois, une lettre est dupliquée (insertion), parfois, deux lettres sont permutées et parfois, l’erreur concerne toute une section. Le résultat, c’est ce que les scientifiques appellent une mutation. Et cette mutation est transmise à la prochaine génération.
Au fil du temps, de plus en plus de mutations s'accumulent dans le virus. Si certaines d'entre elles sont avantageuses pour la propagation, les virus possédant ce matériel génétique s’imposent face aux formes n’ayant pas muté. Un nouveau variant voit le jour. Mais ce n'est pas tout. Au fil du temps, le virus peut muter à un tel point que son mode d'action diffère sensiblement de celui de l'agent pathogène initial. Les chercheurs parlent alors d'une nouvelle souche.
Les virus à ARN tels que le SARS-CoV-2 mutent en règle générale plus fréquemment que les virus à ADN, tels que le virus de l'hépatite B. Mais, contrairement à d'autres virus à ARN comme le virus de l’influenza de type B qui cause la grippe ou le virus de l'immunodéficience humaine (VIH) qui cause le sida, le taux de mutation des coronavirus est beaucoup plus lent. Cela s’explique par le fait que le SARS-CoV-2 et les virus dérivés intègrent un mécanisme de réparation qui corrige du moins partiellement les erreurs de copie.
Quels variants du SARS-CoV-2 font actuellement les grands titres de l’actualité ?
En cette fin d’hiver 2021, trois variants du SARS-CoV-2 font la une de l’actualité : les variants britannique, sud-africain et brésilien. Le variant britannique a la dénomination scientifique B.1.1.7, le variant sud-africain est appelé B.1.351 et le variant brésilien P.1. Les chercheurs américains estiment qu’un quatrième variant est responsable de l’explosion du nombre de cas dans l'État américain de Californie depuis décembre 2020. Le variant connu sous le nom CAL.20C a été décrit dès juillet 2020, mais n'a pas eu beaucoup d’incidence jusqu'en octobre. Ce n'est qu'à la fin de l'année qu'il a commencé à se propager rapidement (Source : Zhang, Wenjuan ; Davis, Brian D. ; Chen, Stephanie S. ; Martinez, Jorge M. Sincuir ; Plummer, Jasmine T. ; Vail, Eric (2021) : Emergence of a novel SARS-CoV-2 strain in Southern California, USA).
Les noms « variant britannique », « variant sud-africain », etc. n'indiquent pas avec certitude leur origine. Ils indiquent le lieu où les variants ont été identifiés pour la première fois.
Les chercheurs s’attendent à ce que de nouvelles mutations continuent d’apparaître, qui s’imposeront face aux mutations existantes ou en supplanteront d'autres.
Que sait-on du variant britannique ?
En décembre 2020, le nombre d'infections a explosé au Royaume-Uni. La cause en était un nouveau variant du virus SARS-CoV-2, qui a été dénommé dans un premier temps VUI-202012/01 (« V » pour « Variant », « U » pour « under » et « I » pour « investigation »). Ensuite, il a également été appelé VOC-202012/01 (pour « Variant Of Concern », littéralement « variant préoccupant »). Il est mieux connu sous la désignation B.1.1.7.
Les mutations les plus importantes du variant britannique comprennent pas moins de quatre modifications dans la partie du génome qui contient la notice de synthèse de la protéine Spike.
Les pointes de la couronne
Par « spike », on désigne les « pointes de la couronne d'un coronavirus ». Ces minuscules pointes à sa surface permettent au virus de pénétrer dans la cellule hôte. Cependant, elles sont également le lieu d’action du système immunitaire contre le virus. Ainsi, l’organisme fabrique des anticorps qui s’agrippent aux pointes et empêchent le virus de pénétrer dans la cellule, si bien que ce dernier est neutralisé.
Deux mutations du variant britannique – appelées N501Y et K417N – renforcent la liaison entre le virus et la cellule hôte. Une autre mutation, appelée E484K, a modifié la protéine Spike de sorte que certains anticorps ne peuvent plus la reconnaître et s’y agripper.
Ce sont ces modifications qui auraient permis au variant britannique de se propager plus facilement. De premiers modèles mathématiques sont partis du principe qu'il était entre 50 et 74 % plus contagieux. Des estimations plus récentes de l'autorité de santé britannique suggèrent une hausse de la contagiosité de l'ordre de 25 à 40 %. La Task Force responsable des projections statistiques en rapport avec la pandémie de Covid-19 a estimé que la contagiosité avait augmenté de 39 %. Dans ses modèles, l'apprentissage machine, un outil issu du domaine de l'intelligence artificielle, aide la Task Force à dégager des tendances dans les données de la Direction de la Santé. Selon Joël Mossong, épidémiologiste à l'Inspection sanitaire, les données du Contact Tracing au Luxembourg suggèrent que le variant britannique au Luxembourg est jusqu'à 70 % plus contagieux que les variants qui circulaient jusqu’à présent, car la probabilité que les contacts soient testés positifs a augmenté. Une certaine incertitude demeure donc à ce sujet. C’est pourquoi, pour évoquer toutes ces incertitudes, Joël Mossong pense que le variant britannique est probablement 30 à 80 % plus contagieux.
Selon les conclusions préliminaires, les vaccins développés et autorisés à ce jour devraient également s’avérer efficaces contre le variant britannique.
Au Luxembourg, ce variant est devenu prédominant. Dans un échantillon représentatif, le variant britannique représentait 57,6% des cas positifs. Pour cet échantillon représentatif, 132 échantillons ont été séquencés, ce qui représente 12 % de l’ensemble des cas positifs de cette semaine. Le Centre européen de prévention et de contrôle des maladies (ECDC) recommande un taux de 10 % pour disposer d'un échantillon représentatif.
Que sait-on du variant sud-africain ?
Un nouveau variant du virus a également fait son apparition en Afrique du Sud en décembre 2020. Il est connu sous le nom B.1.351 ou 20H/501Y.V2. Ici aussi, des mutations – N501Y comme pour le variant britannique et K417N – permettent au virus de mieux s’agripper à la cellule hôte. On y retrouve aussi la mutation E484K, qui protège le virus contre certains anticorps. Le problème avec ce variant, c’est qu’il existe des éléments attestant que les vaccins testés jusqu’à présent seraient moins efficaces. Cela concerne plus particulièrement le vaccin de la société pharmaceutique britanno-suédoise AstraZeneca. Dans une étude sud-africaine, le vaccin AZD1222 affichait une efficacité de seulement 21,9 %. Lors de cette étude, 19 des 750 participants ont contracté le virus malgré le fait qu'ils avaient été vaccinés. Chez les 717 sujets qui ont reçu le placebo, 23 infections se sont produites. Cependant, toutes les personnes contaminées ont développé des formes légères à modérées de la maladie. L’étude n'a pas permis de déterminer si le vaccin d'AstraZeneca a une incidence sur le risque de contracter une forme grave de la maladie. On examine encore à l’heure actuelle si le variant sud-africain est également plus contagieux.
Dans l’échantillon représentatif au Luxembourg, le variant sud-africain représentait 4,5 % des cas positifs.
Que sait-on du variant brésilien ?
Le variant détecté au Brésil à la fin de l'année 2020 porte le nom 20J/501Y.V3 ou P.1. Ici aussi, la notice de synthèse de la protéine Spike a été modifiée, ce qui a rendu le virus plus contagieux et plus résistant au système immunitaire de la personne infectée. Ce variant a fait l'objet d'une attention particulière, car il a provoqué une deuxième vague dans la ville brésilienne de Manaus. Après une première vague particulièrement virulente, les chercheurs avaient estimé que 76 % des habitants possédaient des anticorps. On pensait donc que l'immunité collective avait été acquise et qu'il n’allait pas y avoir de deuxième vague. Mais une deuxième vague a bel et bien éclaté en raison du variant brésilien.
Infobox
Manaus, la capitale de l'État brésilien de l'Amazonas, avait déjà beaucoup fait parler d’elle lors de la première vague de Covid-19 en avril 2020. À l'époque, le virus s'était propagé si rapidement que le système de santé de la métropole s'était effondré. Après la fin de la première vague, les tests ont montré que 76 % des habitants avaient fabriqué des anticorps. Plus de 75 % de la population avait donc été en contact avec le virus et devaient être immunisés contre une nouvelle infection, du moins pendant un certain temps. De plus, selon la thèse des épidémiologistes, l’immunité collective avait été atteinte. Le principe de l'immunité collective veut qu’un grand nombre de personnes ne soient pas sensibles à un agent pathogène, ce qui rend sa propagation très difficile, voire impossible. En fonction de l'agent pathogène, ce stade est atteint lorsque 65 à 70 % de la population est vaccinée. Dans le cas de virus particulièrement contagieux, il est possible qu’une proportion nettement plus élevée de personnes doive être immunisée au sein de la population.
Contrairement à l'hypothèse selon laquelle l'immunité collective avait été atteinte, une deuxième vague a déferlé sur Manaus à la fin de l'année 2020, surchargeant une fois de plus le système de santé. Selon une théorie, le nouveau variant pourrait être à l'origine de cette deuxième vague. D'une part, les chercheurs partent du principe que les virus modifiés sont mieux à même d'échapper aux anticorps du système immunitaire. D'autre part, un taux d'infection plus élevé pourrait également avoir eu pour conséquence que le seuil d'immunité collective n'avait pas encore pu être atteint. Et une dernière thèse part du principe qu'en réalité, la proportion de personnes qui possédait des anticorps avait peut-être été surestimée. Cela pourrait être dû aux estimations avancées par les chercheurs à partir d’échantillons pour déterminer le nombre total de personnes possédant des anticorps.
Qu'est-ce qui est pire : un variant plus contagieux ou un variant plus mortel ?
La dangerosité d'un virus dépend de plusieurs facteurs. Deux d'entre eux sont la virulence (c’est-à-dire le caractère infectieux du virus) et la létalité (sa mortalité).
Si un variant s’accompagne d’une mortalité accrue – une hypothèse qui est actuellement envisagée pour le variant britannique, mais pas encore prouvée – cela augmente le risque de succomber à l'infection. Ainsi, une plus grande létalité rend le virus plus dangereux pour la personne concernée. Un variant plus contagieux, en revanche, a pour conséquence que davantage de personnes sont infectées en même temps. D’un point de vue purement statistique, une plus grande contagiosité s’accompagne bien entendu aussi d'une hausse du nombre de décès. Davantage de personnes doivent être hospitalisées, ce qui pourrait à terme pousser le système de santé au point de rupture. À partir du moment où un traitement adéquat des cas graves n'est plus possible en raison d’une pénurie de respirateurs ou d'oxygène, le nombre de décès augmente également. Selon l'épidémiologiste Adam Kucharski, une virulence plus élevée pourrait par conséquent causer plus de décès qu’une plus grande létalité, et rend donc le virus plus dangereux, notamment pour la société.
En outre, d'autres facteurs ont une influence à cet égard. Ainsi, certains scientifiques pensent que le variant britannique prolonge aussi la durée de la maladie, ce qui étendrait la période pendant laquelle un patient atteint de Covid-19 est contagieux. Dans cette situation, le système de santé se verrait confronté aux mêmes défis qu’avec une contagiosité plus élevée. D'autre part, on pense que les nouveaux variants s’accompagnent généralement d’une évolution plus grave de la maladie, si bien que le nombre de cas asymptomatiques – c'est-à-dire de personnes présentant une forme plus légère et discrète de la maladie – diminuerait. Cet effet permettrait, à son tour, de mieux contenir la pandémie, car moins il y a de formes asymptomatiques ou peu symptomatiques de la maladie, moins il y a de chaînes d'infection qui passent inaperçues et meilleur sera le suivi.
Quel effet ces variants ont-ils sur le déroulement de la pandémie ?
De manière générale, les scientifiques estiment que les nouveaux variants pourraient à nouveau aggraver la pandémie. En effet, le nouveau variant a proliféré au Royaume-Uni malgré les mesures de confinement. Selon les estimations actuelles des scientifiques, le taux de reproduction – c'est-à-dire le nombre de personnes qu'un individu infecté contamine en moyenne – s'élève actuellement à 1,4. La forme du virus qui circulait jusqu’à présent est associée actuellement à un taux de reproduction de 0,9. Un variant plus contagieux a aussi pour conséquence de retarder le moment où l'immunité collective est atteinte, c'est-à-dire le moment où un nombre plus important de personnes sont immunisées après avoir guéri de l’infection ou par vaccination.
Au Luxembourg, la Task Force responsable des projections statistiques travaille sur les prévisions concernant l’évolution de la pandémie. Les scientifiques utilisent un modèle qui projette l'évolution de la pandémie à partir de données actuelles relatives aux infections en appliquant des méthodes issues de l’apprentissage machine. Si l'on fait abstraction du variant britannique, le modèle prévoit une nouvelle baisse du nombre de cas pour la seconde moitié du mois de février. Si l'on tient compte du variant britannique, un tournant se dessine toutefois à la fin du mois de février, car le nombre de cas augmenterait à nouveau à partir de ce moment-là. Si les interactions sociales se poursuivent aux niveaux actuels, les chercheurs s'attendent à ce que le prochain pic épidémique se produise en mai. Selon les prévisions, il atteindrait le même niveau que celui d’octobre 2020. Avec près de 600 cas par jour pour le Luxembourg, le système de santé avait presque atteint son seuil de saturation à l'époque.
Toutefois, ces modèles partent du principe que le comportement de la société reste inchangé. Or, lorsque la situation devient critique, les gens adaptent généralement leur comportement – ou les responsables politiques prennent de nouvelles mesures. Les modélisations à moyen ou long terme s’accompagnent donc de grandes incertitudes. Ils illustrent ce qui se passerait si la tendance actuelle se poursuivait. En d'autres termes, elles ne font pas office d’outil de prévision, mais elles indiquent la tendance pour les jours et les semaines à venir si la tendance du moment se poursuit. Il s’agit de valeurs indicatives, mais pas de prédictions. C'est là que des malentendus surviennent souvent dans le débat public.
Qu'est-ce qui favorise les mutations et pourrait-on les arrêter ?
Comme nous l’avons déjà évoqué au début de l'article, les mutations sont des phénomènes aléatoires qui n'ont pas nécessairement d'effet sur le virus et sa fonction. Elles se produisent avec une certaine probabilité dès que le virus est répliqué dans la cellule hôte. Cela signifie que plus le nombre de personnes infectées par le virus est élevé, plus ce dernier est copié souvent et plus des erreurs risquent de se produire dans la notice de synthèse. Si certaines d'entre elles sont avantageuses pour la propagation, les virus possédant ce matériel génétique muté s’imposent face aux virus non mutés.
Tout ce qui favorise la propagation du virus est bénéfique dans ce contexte : une plus grande contagiosité, une meilleure dissimulation face au système immunitaire, etc. La fréquence à laquelle de nouveaux variants s'imposent et la direction dans laquelle ils évoluent dépendent de leur environnement. Dans le corps, il s'agit principalement du système immunitaire, qui a pour tâche d'éliminer rapidement le virus. C’est pourquoi les variants apparus de manière fortuite qui échappent aux attaques du système immunitaire sont plus susceptibles de se répandre et de remplacer les formes initiales. Cela pourrait entraîner l'émergence de nouveaux variants qui échappent au moins à certaines attaques du système immunitaire. Cependant, le système immunitaire possède plusieurs armes et s'adapte à son tour aux virus modifiés.
Il n'est pas possible de prévenir entièrement les mutations. Et l'idée d'empêcher certains variants de se propager s’est avérée impossible à mettre en œuvre.
Quelle est la probabilité que des variants plus graves fassent leur apparition ou, au contraire, que le virus s'affaiblisse ?
Dans l'état actuel des connaissances, le virus SARS-CoV-2 est une zoonose. C'est ainsi que les scientifiques désignent un agent pathogène normalement présent chez les animaux qui a été transmis accidentellement à l'homme. Dans le cas du virus SARS-CoV-2, les chauves-souris sont considérées comme les hôtes les plus probables du virus. Les animaux qui sont confrontés depuis longtemps à un tel agent pathogène ont développé une certaine coexistence. Bien qu'ils soient porteurs du virus, ils ne tombent généralement pas malades.
Si l'agent pathogène passe à l’être humain, la situation est différente. Pour le virus, nous sommes un territoire étranger et notre système immunitaire n’est pas familiarisé avec le virus. La situation peut se détériorer rapidement. Les nouvelles zoonoses entraînent donc souvent des évolutions graves de la maladie et sont plus susceptibles d'être fatales. Cela est parfois aussi dû au fait que le système immunitaire surréagit et provoque de graves réactions inflammatoires dans l'organisme – comme c'est le cas avec la Covid-19. Avec le temps, cependant, l'intrus et le nouvel hôte apprennent à mieux se connaître. Le système immunitaire affûte ses armes et les variants du virus qui lui échappent persistent. Le virus devient local, mais il cause en règle générale moins de dommages à son hôte.
En effet, au début de la pandémie déjà , on avait constaté l'existence de variants s’accompagnant d’une évolution plus favorable de la maladie. Les scientifiques ont toutefois souligné à l'époque qu'il ne s'agissait que de variants locaux. Jusqu'à présent, ces variants n'ont pas repoussé la forme prédominante du virus. (sources 22 et 23 dans l'infobox)
Auteurs : Kai Dürfeld (scienceRELATIONS - Communication scientifique), Jean-Paul Bertemes (FNR)
Infobox
- Roossinck, Marilyn J. (2020): Viren! Helfer, Feinde, Lebenskünstler - in 101 Porträts. 2. Auflage 2020. Berlin: Springer Berlin; Springer.
- Corum, Jonathan; Zimmer, Carl (2021): Coronavirus Variants and Mutations. The New York Times. Online verfügbar unter https://www.nytimes.com/interactive/2021/health/coronavirus-variant-tracker.html?campaign_id=34&emc=edit_sc_20210209&instance_id=26918&nl=science-times®i_id=75221209&segment_id=51277&te=1&user_id=7dfb557d6b820bd9edaad4dca10d0931, zuletzt aktualisiert am 15.02.2021, zuletzt geprüft am 17.02.2021.
- Sanjuán, Rafael; Nebot, Miguel R.; Chirico, Nicola; Mansky, Louis M.; Belshaw, Robert (2010): Viral mutation rates. In: Journal of virology 84 (19), S. 9733–9748. DOI: 10.1128/JVI.00694-10.
- Thal, Dana (2021): Mutationen – ein Tauziehen zwischen Virus und Wirt. Nationale Forschungsplattform für Zoonosen. Online verfügbar unter https://zoonosen.net/mutationen-ein-tauziehen-zwischen-virus-und-wirt, zuletzt aktualisiert am 17.02.2021, zuletzt geprüft am 21.02.2021.
- Zhang, Wenjuan; Davis, Brian D.; Chen, Stephanie S.; Martinez, Jorge M. Sincuir; Plummer, Jasmine T.; Vail, Eric (2021): Emergence of a novel SARS-CoV-2 strain in Southern California, USA.
- Mölling, Karin (2020): Viren. Supermacht des Lebens. 1. Auflage. München: C.H.Beck.
- Volz, Erik; Mishra, Swapnil; Chand, Meera; Barrett, Jeffrey C.; Johnson, Robert; Geidelberg, Lily et al. (2021): Transmission of SARS-CoV-2 Lineage B.1.1.7 in England: Insights from linking epidemiological and genetic data. DOI: 10.1101/2020.12.30.20249034.
- Davies, Nicholas G.; Abbott, Sam; Barnard, Rosanna C.; Jarvis, Christopher I.; Kucharski, Adam J.; Munday, James et al. (2020): Estimated transmissibility and severity of novel SARS-CoV-2 Variant of Concern 202012/01 in England. In: medRxiv, 2020.12.24.20248822. DOI: 10.1101/2020.12.24.20248822.
- Public Health England (2021): Investigation of novel SARS-CoV-2 variant. Variant of Concern 202012/01. Technical briefing 5. Public Health England. Online verfügbar unter https://assets.publishing.service.gov.uk/government/uploads/system/uploads/attachment_data/file/959426/Variant_of_Concern_VOC_202012_01_Technical_Briefing_5.pdf, zuletzt geprüft am 22.02.2021.
- Tertilt, Mathias; Gerhard, Saskia (2021): Corona: Wie gefährlich sind die neuen Mutationen? Neue Virusvarianten. Online verfügbar unter https://www.quarks.de/gesundheit/medizin/corona-wie-gefaehrlich-sind-die-neuen-mutationen/, zuletzt aktualisiert am 26.01.2021, zuletzt geprüft am 08.02.2021.
- Greaney, Allison J.; Loes, Andrea N.; Crawford, Katharine H.D.; Starr, Tyler N.; Malone, Keara D.; Chu, Helen Y.; Bloom, Jesse D. (2021): Comprehensive mapping of mutations to the SARS-CoV-2 receptor-binding domain that affect recognition by polyclonal human serum antibodies. DOI: 10.1101/2020.12.31.425021.
- Wang, Zijun; Schmidt, Fabian; Weisblum, Yiska; Muecksch, Frauke; Barnes, Christopher O.; Finkin, Shlomo et al. (2021): mRNA vaccine-elicited antibodies to SARS-CoV-2 and circulating variants. In: bioRxiv : the preprint server for biology. DOI: 10.1101/2021.01.15.426911.
- Madhi, Shabir A.; Baillie, Vicky; Cutland, Clare L.; Voysey, Merryn; Koen, Anthonet L.; Fairlie, Lee et al. (2021): Safety and efficacy of the ChAdOx1 nCoV-19 (AZD1222) Covid-19 vaccine against the B.1.351 variant in South Africa. DOI: 10.1101/2021.02.10.21251247.
- Tegally, Houriiyah; Wilkinson, Eduan; Giovanetti, Marta; Iranzadeh, Arash; Fonseca, Vagner; Giandhari, Jennifer et al. (2020): Emergence and rapid spread of a new severe acute respiratory syndrome-related coronavirus 2 (SARS-CoV-2) lineage with multiple spike mutations in South Africa. DOI: 10.1101/2020.12.21.20248640.
- Buss, Lewis F.; Prete, Carlos A.; Abrahim, Claudia M. M.; Mendrone, Alfredo; Salomon, Tassila; Almeida-Neto, Cesar de et al. (2021): Three-quarters attack rate of SARS-CoV-2 in the Brazilian Amazon during a largely unmitigated epidemic. In: Science (New York, N.Y.) 371 (6526), S. 288–292. DOI: 10.1126/science.abe9728.
- Fine, Paul; Eames, Ken; Heymann, David L. (2011): "Herd immunity": a rough guide. In: Clinical infectious diseases : an official publication of the Infectious Diseases Society of America 52 (7), S. 911–916. DOI: 10.1093/cid/cir007.
- Sabino, Ester C.; Buss, Lewis F.; Carvalho, Maria P. S.; Prete, Carlos A.; Crispim, Myuki A. E.; Fraiji, Nelson A. et al. (2021): Resurgence of COVID-19 in Manaus, Brazil, despite high seroprevalence. In: The Lancet 397 (10273), S. 452–455. DOI: 10.1016/S0140-6736(21)00183-5.
- Faria, Nuno R. (2021): Genomic characterisation of an emergent SARS-CoV-2 lineage in Manaus: preliminary findings. Online verfügbar unter https://virological.org/t/genomic-characterisation-of-an-emergent-sars-cov-2-lineage-in-manaus-preliminary-findings/586, zuletzt aktualisiert am 12.01.2021, zuletzt geprüft am 21.02.2021.
- https://twitter.com/adamjkucharski/status/1343567425107881986?lang=de
- Hintergrundgespräch mit Dr. Alexander Skupin, Associate professor/Chief scientist 2 in Modelling of Biomedical Data am LCSB der Universität Luxemburg und Leiter der Task-Force-Gruppe für die statistischen Projektionen in der Covid-19 Pandemie
- Sun, Xinjuan; Wang, Tianyuan; Cai, Dayong; Hu, Zhiwei; Chen, Jin’an; Liao, Hui et al. (2020): Cytokine storm intervention in the early stages of COVID-19 pneumonia. In: Cytokine & Growth Factor Reviews 53, S. 38–42. DOI: 10.1016/j.cytogfr.2020.04.002.
- Toyoshima, Yujiro; Nemoto, Kensaku; Matsumoto, Saki; Nakamura, Yusuke; Kiyotani, Kazuma (2020): SARS-CoV-2 genomic variations associated with mortality rate of COVID-19. In: Journal of human genetics 65 (12), S. 1075–1082. DOI: 10.1038/s10038-020-0808-9.
- Young, Barnaby E.; Fong, Siew-Wai; Chan, Yi-Hao; Mak, Tze-Minn; Ang, Li Wei; Anderson, Danielle E. et al. (2020): Effects of a major deletion in the SARS-CoV-2 genome on the severity of infection and the inflammatory response: an observational cohort study. In: The Lancet 396 (10251), S. 603–611. DOI: 10.1016/S0140-6736(20)31757-8.